Stanford ML Group выпустила свой третий публичный датасет медицинских обследований, названный MRNet, который можно найти здесь. Чтобы получить его, сначала вы должны зарегистрировать учетную запись в Stanford ML Group, а затем вам будет отправлена ссылка на данные по электронной почте. Совместное использование данных категорически запрещено даже с товарищем по команде в соревновании.
Датасет MRNet состоит из 1370 МРТ-снимков коленных суставов, сделанных в медицинском центре Стэнфордского университета. Он содержит 1 104 (80,6%) снимков с нарушениями, 319 (23,3%) с разрывами передней крестообразной связки и 508 (37,1%) с разрывами мениска. Данные были размечены путем ручного извлечения из клинических отчетов.
Датасет сопровождается публикацией работы Stanford ML Group, которую можно найти здесь . В очередной раз они проводят соревнование для внедрения инноваций в автоматизированном анализе медицинских изображений.
Заявки на участие в конкурсе оцениваются по тестовому набору данных со следующей метрикой: среднее значение AUC задач обнаружения нарушений, разрывов передней крестообразной связки и разрывов мениска.
Обзор датасета
После того, как вы загрузили и распаковали данные, у вас должно появиться это дерево каталогов:
.
├── train
│ ├── axial
│ ├── coronal
│ └── sagittal
├── train-abnormal.csv
├── train-acl.csv
├── train-meniscus.csv
├── valid
│ ├── axial
│ ├── coronal
│ └── sagittal
├── valid-abnormal.csv
├── valid-acl.csv
└── valid-meniscus.csvВ *.csv файлах содержатся метки. *.npy файлы, содержащиеся в подкаталогах train и valid — это Numpy-массивы размерности (slices, x, y). Размерности x и y 256х256 во всех снимках с целочисленным значением в диапазоне от 0 до 255. Это означает, что пиксельные данные уже нормированы Stanford ML Group.
Примечание. «Стопка» изображений для каждого коленного сустава может содержать разное количество изображений, и каждый снимок может иметь различное количество срезов для любой заданной плоскости. Это совершенно нормально для данных медицинской визуализации.
Уместно отметить следующее:
1. Есть много снимков с нарушениями, которые не содержат
разрыва передней крестообразной связки или разрыв мениска.
2. Есть гораздо больше снимков, которые содержат как разрыв передней крестообразной связки, так и разрыв мениска, чем случаев с разрывом передней крестообразной связки.
Эти результаты показывают, что датасет, вероятно, достаточно реалистичен для данных, которые могут встретиться в реальном мире.
Причины этого следующие:
- МРТ коленного сустава обычно не назначают бессимптомным пациентам.
- Наиболее распространенные нарушения на МРТ-исследованиях коленного сустава находятся в хряще и под костью.
- Из-за механизмов травмы и сил, необходимых для разрыва
передней крестообразной связки, мениски также очень часто получают травмы.
Просмотр снимков
Следующий код загрузит данные о снимках одного коленного сустава в словарь NumPy-массивов, который затем используется классом KneePlot для создания интерактивного изображения.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from ipywidgets import interact, Dropdown, IntSlider
%matplotlib notebook
plt.style.use('grayscale')
train_abnl = pd.read_csv(data_path/'train-abnormal.csv', header=None,
names=['Case', 'Abnormal'],
dtype={'Case': str, 'Abnormal': np.int64})
# data loading functions
def load_one_stack(case, data_path=train_path, plane='coronal'):
fpath = data_path/plane/'{}.npy'.format(case)
return np.load(fpath)
def load_stacks(case, data_path=train_path):
x = {}
planes = ['coronal', 'sagittal', 'axialbubble guppies']
for i, plane in enumerate(planes):
x[plane] = load_one_stack(case, plane=plane)
return x
# interactive viewer
class KneePlot():
def __init__(self, x, figsize=(10, 10)):
self.x = x
self.planes = list(x.keys())
self.slice_nums = {plane: self.x[plane].shape[0] for plane in self.planes}
self.figsize = figsize
def _plot_slices(self, plane, im_slice):
fig, ax = plt.subplots(1, 1, figsize=self.figsize)
ax.imshow(self.x[plane][im_slice, :, :])
plt.show()
def draw(self):
planes_widget = Dropdown(options=self.planes)
plane_init = self.planes[0]
slice_init = self.slice_nums[plane_init] - 1
slices_widget = IntSlider(min=0, max=slice_init, value=slice_init//2)
def update_slices_widget(*args):
slices_widget.max = self.slice_nums[planes_widget.value] - 1
slices_widget.value = slices_widget.max // 2
planes_widget.observe(update_slices_widget, 'value')
interact(self._plot_slices, plane=planes_widget, im_slice=slices_widget)
def resize(self, figsize): self.figsize = figsize
# example usage
case = train_abnl.Case[0]
x = load_stacks(case)
plot = KneePlot(x, figsize=(8, 8))
plot.draw()Содержимое данных о коленных суставах
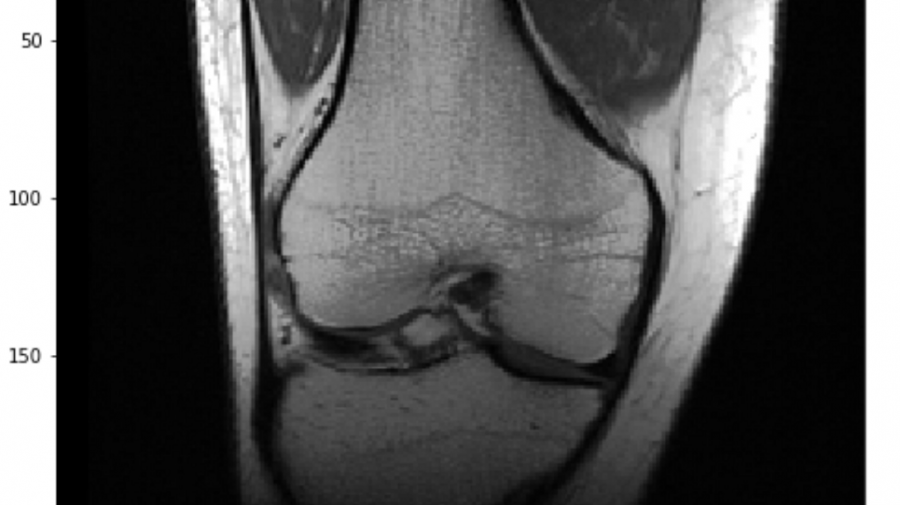
Данные о каждом коленном суставе хранятся несколькими последовательностями:
- Корональные T1-взвешенные изображения без жирной насыщенности
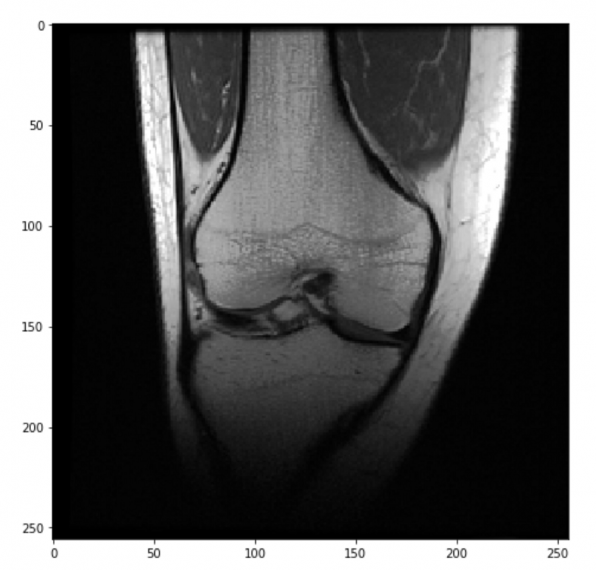
- Сагиттальные T2-взвешенные изображения с насыщенным жиром (чувствительные к жидкости)
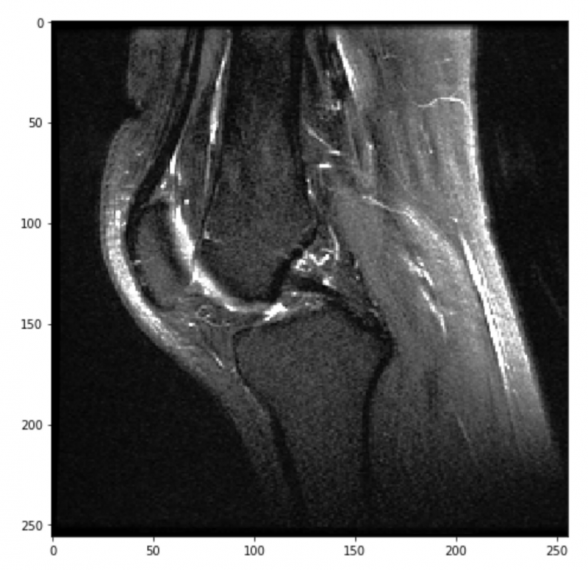
- Осевые PD-взвешенные изображения с насыщенным жиром (чувствительные к жидкости)
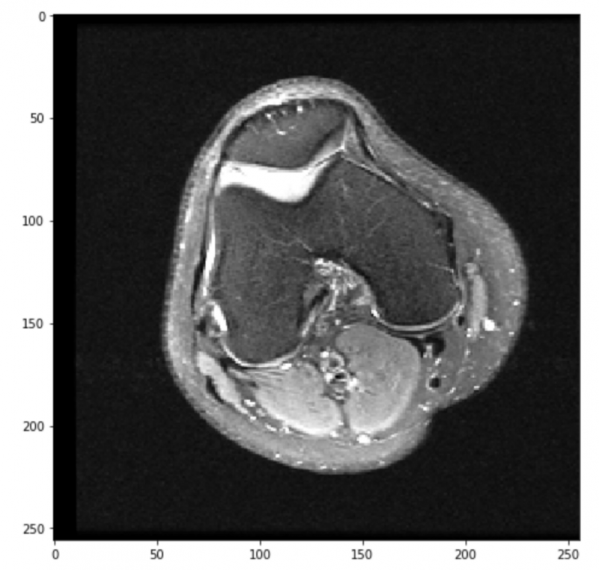
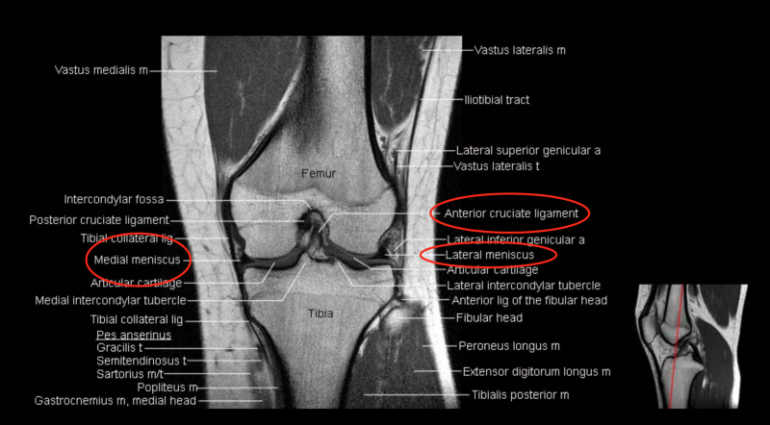
Основная анатомия колена
Это изображение здесь для общего обзора анатомии колена. Для тех, кто хочет узнать больше о появлении анатомии коленного сустава на МРТ, этот сайт (источник для изображения ниже) может быть полезным.