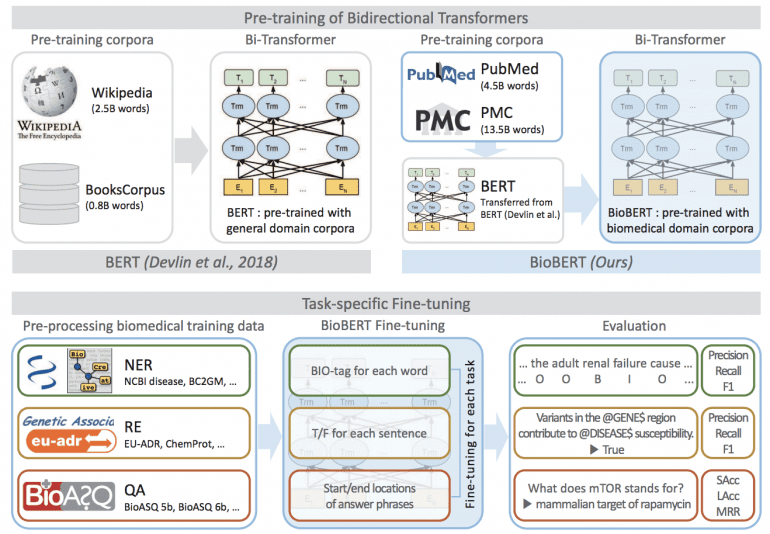
BioBERT — это доработанная модель оригинальной BERT, натренированная для обработки медицинских текстов. Её можно применять для распознавания именованных сущностей (NER), извлечение семантических связей (RE), ответов на вопросы (QA), поиска информации и других задач NLP.
BERT от Google AI была создана для понимания языка общего назначения, BioBERT использует специфичные для предметной области знания из большого набора аннотированных биомедицинских текстов. Доработанная нейросеть превосходит в точности современные аналоги в задачах анализа биомедицинского текста.
Датасеты
Оригинальная BERT обучена на огромных корпусах текстов из Википедии (2,5 миллиарда слов) и BooksCorpus (0,8 миллиарда слов). Ученые Университета Корё и стартапа Clova AI Research дообучили ее на корпусах биомедицинских текстов PubMed и PMC. Они содержат 4,5 миллиарда и 13,5 миллиардов слов соответственно.
Обучение BioBert
BioBERT обучалась более 20 дней, используя 8 графических процессоров V100. Однако для доработки модели (fine-tuning) разработчикам понадобился всего час и видеокарта Titan X. Это связано с небольшим размером набора данных и значительной репрезентативной способностью BioBERT благодаря крупномасштабной предварительной подготовке.
Результаты
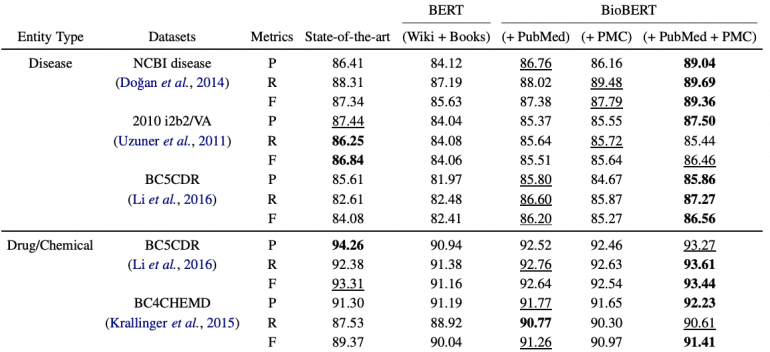
BioBERT превосходит современные модели в задачах анализа биомедицинских текстов. В задаче выделения именованных сущностей модель показывает абсолютное улучшение на 0,51%, в извлечении связей — 3,49%, в ответах на вопросы — 9,61%. Подробнее о результатах можно прочитать в статье, посвященной разработке.