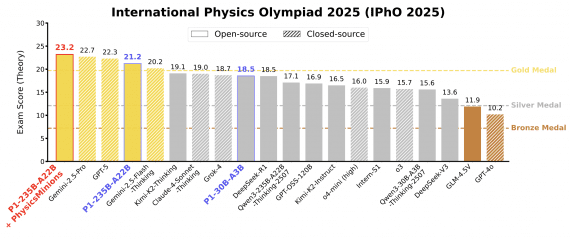
В последние годы был достигнут значительный прогресс в разработке как специализированных, так и универсальных моделей сегментации 2D медицинских изображений, однако область 3D и видеосегментации остается недостаточно исследованной. Группа исследователей из университетов Канады и Гарварда, США, представила MedSAM2 — модель для сегментации медицинских изображений и видео в 3D, построенная на базе модели SAM2.1-Tiny. MedSAM2 — полностью открытая модель: исследователи выложили код, веса модели и аннотированные датасеты на Github. Исследователям удалось сократить время разметки изображений на 85% благодаря использования метода Human-in-the-loop.
Архитектура модели
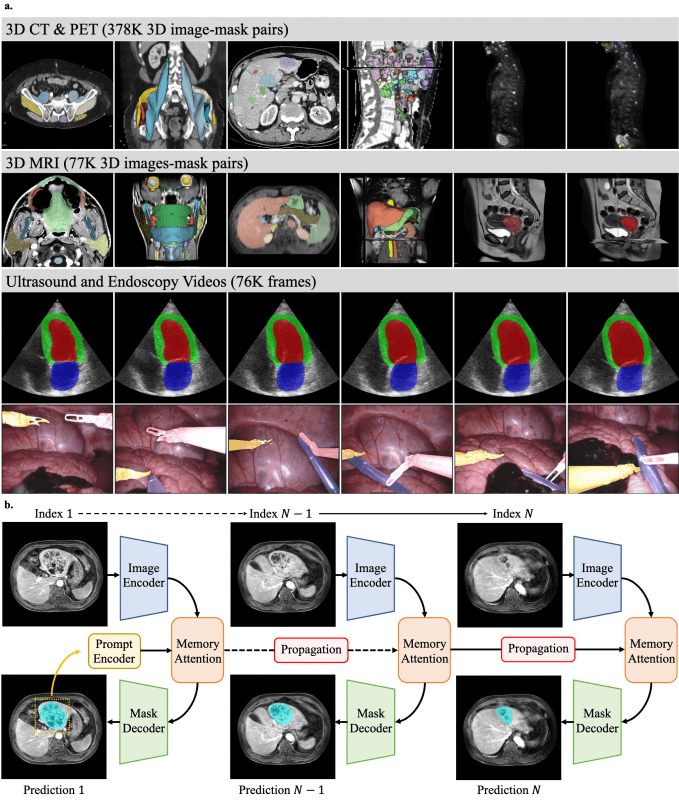
MedSAM2 заметно лучше конкурентов благодаря нескольким ключевым инновациям:
- Оптимизация архитектуры: Построена на основе SAM2 с модификациями для специфики медицинской области, особенно для обработки 3D данных. Модель использует иерархический трансформер (Hiera) с модулем внимания памяти, который обрабатывает 3D-сканы или кадры из видео.
- Комплексный набор данных для обучения: дообучена на объемном наборе данных, содержащем более 455,000 пар 3D изображение-маска для КТ (363,161), ПЭТ (14,818) и МРТ (77,154), плюс 76,000 видеокадров ультразвука и эндоскопии.
- Обработка с памятью: использует потоковый «банк» памяти, который обусловливает признаки текущего кадра на основе предсказаний предыдущих кадров, обеспечивая эффективное распространение масок сегментации по 3D объемам или последовательным видеокадрам.
- Подход с transfer-learning: модель достигает высокой производительности благодаря дообучению легковесного варианта SAM2.1-Tiny, с дифференцированными скоростями обучения — ниже для энкодера изображений, чтобы сохранить предварительно обученные возможности извлечения признаков, и выше для других компонентов, чтобы адаптироваться к характеристикам медицинской области.
Результаты и сравнения
MedSAM2 демонстрирует значительные улучшения по сравнению с существующими моделями сегментации в комплексных оценках. Ниже приведены сравнительные показатели производительности MedSAM2 в сравнении с вариантами SAM2.1 (Tiny, Small, Base, Large) и EfficientMedSAM-Top1:
Сегментация 3D изображений (коэффициент сходства Дайса)
| Задача | MedSAM2 | EfficientMedSAM-Top1 | Лучший вариант SAM2.1 |
|---|---|---|---|
| Органы КТ | 88.84% (80.03-94.03%) | 83.55% (67.20-91.78%) | ~80% (различные модели) |
| Поражения КТ | 86.68% (74.32-91.14%) | 77.95% (69.15-84.81%) | ~70% (различные модели) |
| Органы МРТ | 87.06% (82.96-90.04%) | 74.83% | ~84% (SAM2.1-Base) |
| Поражения МРТ | 88.37% (79.91-93.26%) | 82.25% (68.30-90.53%) | ~72% (различные модели) |
| Поражения ПЭТ | 87.22% (79.07-90.45%) | 77.85% | ~80% (SAM2.1-Large) |
Результаты:
- MedSAM2 превосходит EfficientMedSAM-Top1 на 5-13% в зависимости от задачи, SAM2.1 на 7-18%;
- Все варианты SAM2.1 показали схожую производительность независимо от размера модели (нет статистически значимых различий, p>0.05);
- Разрыв в производительности особенно заметен для поражений, которые традиционно сложнее сегментировать.
Сегментация видео (коэффициент сходства Дайса)
ЛЖ — левый желудочек сердца, ЛП — левое предсердие.
| Задача | MedSAM2 | SAM2.1-Tiny | SAM2.1-Small | SAM2.1-Base | SAM2.1-Large |
|---|---|---|---|---|---|
| УЗИ ЛЖ | 96.13% (95.09-97.15%) | ~94% | ~94% | ~94% | ~94% |
| УЗИ эпикард ЛЖ | 93.10% (91.07-94.11%) | ~88% | ~89% | ~89% | ~90% |
| УЗИ ЛП | 95.79% (94.38-96.96%) | ~93% | ~93% | ~94% | ~94% |
| Полипы эндоскопии (легкие) | 92.24% (85.15-96.11%) | 92.11% (75.74-96.47%) | 93.32% (76.24-96.58%) | 93.87% (77.48-96.64%) | 93.76% (77.20-96.60%) |
| Полипы эндоскопии (сложные) | 92.22% (83.37-95.88%) | 83.43% (60.34-92.53%) | 84.93% (63.32-92.87%) | 85.64% (64.55-92.98%) | 87.47% (67.21-93.51%) |
Значимые результаты:
- Для УЗИ сердца MedSAM2 показывает улучшение на 2-4% по сравнению с вариантами SAM2.1;
- Для сложных случаев полипов MedSAM2 поддерживает стабильную производительность с улучшением на 5-9% по сравнению с моделями SAM2.1;
- MedSAM2 демонстрирует значительно меньшую вариабельность (более узкие межквартильные диапазоны) во всех задачах, что указывает на более надежную и стабильную сегментацию
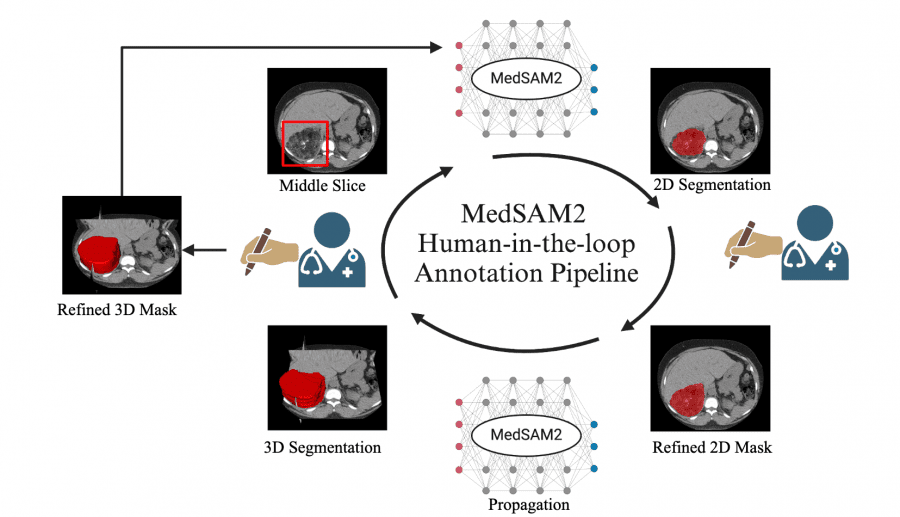
Пайплайн аннотации с человеком в контуре
Возможно, наиболее значительный вклад MedSAM2 – это ее практическое применение в рабочих процессах аннотации данных. Исследователи реализовали пайплайн аннотации с человеком в цикле (human-in-the-loop), который значительно сокращает время ручной аннотации:
- Аннотация поражений КТ: Сократилось время аннотации с 525.9 секунд до 74.3 секунд на поражение (на 85.86% быстрее) за три итеративных раунда, что позволило аннотировать 5,000 поражений КТ;
- Аннотация поражений печени на МРТ: уменьшилось время аннотации с 520.3 секунд до 65.2 секунд на поражение (на 87.47% быстрее), что ускорило аннотацию 3,984 поражений печени на МРТ;
- Аннотация видео эхокардиографии: cократилось время аннотации с 102.3 секунд до 8.4 секунд на кадр (на 91.79% быстрее), что позволило аннотировать 251,550 кадров из 1,800 видео.
Этот итеративный процесс демонстрирует, как производительность модели улучшается с каждым раундом обратной связи от человека, создавая благотворный цикл, который постоянно повышает эффективность аннотации.
Доступность и развертывание с открытым исходным кодом
Ключевым преимуществом MedSAM2 является ее полностью открытый исходный код, предоставляющий исследовательскому и клиническому сообществу полный доступ к:
- Полному исходному коду: все детали реализации модели и пайплайны обучения;
- Предобученным весам модели: готовые к использованию модели без необходимости дорогостоящего переобучения;
- Аннотированным наборам данных: ценные ресурсы для бенчмаркинга и дальнейших исследований;
- Плагину для 3D Slicer: расширение с открытым исходным кодом для популярной платформы медицинской визуализации.
Технические ограничения
MedSAM2 имеет ряд ограничений:
- Зависимость от boundary box: опора на прямоугольные рамки в качестве основных подсказок ограничивает возможности сегментации сложных анатомических структур, таких как тонкие, разветвленные сосуды.
- Фиксированная конструкция памяти: банк памяти на восемь кадров может быть недостаточным для быстрых или больших движений объекта, потенциально вызывая сбои отслеживания.
- Вычислительные требования: несмотря на использование легковесного варианта SAM2.1-Tiny, вывод по-прежнему требует вычислений на GPU, ограничивая развертывание в ресурсно-ограниченных средах.
Заключение
MedSAM2 представляет собой значительный вклад в анализ медицинских изображений, преодолевая разрыв между общими фундаментальными моделями и специфическими для медицинской области приложениями. Ее способность обрабатывать как 3D снимки, так и видео, в сочетании с существенным повышением эффективности аннотации, делает ее ценным инструментом как для исследований, так и для клинического применения. Значительно снижая время аннотации, MedSAM2 облегчает создание более крупных, высококачественных наборов данных, которые будут дальше продвигать прогресс в медицинском ИИ.
Нельзя переоценить важность открытого исходного кода этого проекта. Предоставляя свободный доступ к коду, весам модели, аннотированным наборам данных и инструментам развертывания, исследователи устранили значительные барьеры для входа в область продвинутой сегментации медицинских изображений.